2020年 | プレスリリース・研究成果
mRNAの安定性を決定する新たな分子機構の発見 〜遺伝子発現の根幹を監視する新たな仕組み〜
【発表のポイント】
- 様々な疾患の原因となるメッセンジャー(伝令)RNA(mRNA)の安定性を決定するコドン(遺伝暗号)の最適度は、mRNAの分解に関与するCcr4-Not複合体によって監視されていることを、遺伝学・生化学的な機能解析によって証明しました。
- クライオ電子顕微鏡を用いた単粒子解析によって、Ccr4-Not複合体とリボソームの結合様式を明らかにしました。
- Ccr4-Not複合体は、発生・細胞分化や癌、炎症に寄与することが報告されており、今回の研究成果は、これまで原因不明とされてきた幅広い疾患の発症機構の解明につながることが期待されます。
【概要】
個々のmRNAがもつ固有の安定性は、コドンの最適度によって調節されていることが報告され、最適度が高いコドンを持つmRNAほど安定であり、最適度が低いコドンを持つmRNAは不安定であるという一般則が確立されています。mRNAの安定性制御は遺伝子発現の根幹であり、その破綻は様々な疾患の原因になります。しかし、コドンの最適度によって調節される翻訳の伸長速度を監視し、個々のmRNAがもつ固有の安定性を決定する機構は不明でした。東北大学大学院薬学研究科の稲田利文教授、松尾芳隆助教とドイツミュンヘン大学Roland Beckmann教授、ケースウェスタンリザーブ大学のJeff Coller教授らの研究グループは、mRNAの安定性を決定する新たな分子機構を発見しました。今回の研究成果は、これまで原因不明とされてきた幅広い疾患の発症機構の解明につながることが期待されます。
本研究成果は、2020年4月17日(金曜日)に米国科学誌『Science』に掲載されました。本研究は、文部科学省科学研究費補助金(基盤研究(A)、(C)、新学術領域研究「ケモテクノロジーが拓くユビキチンニューフロンティア」)、日本医療研究開発機構の革新的先端研究開発支援事業(AMED-CREST)「全ライフコースを対象とした個体の機能低下機構の解明」、(公財)武田科学振興財団、(公財)加藤記念バイオサイエンス振興財団の研究助成により実施しました。
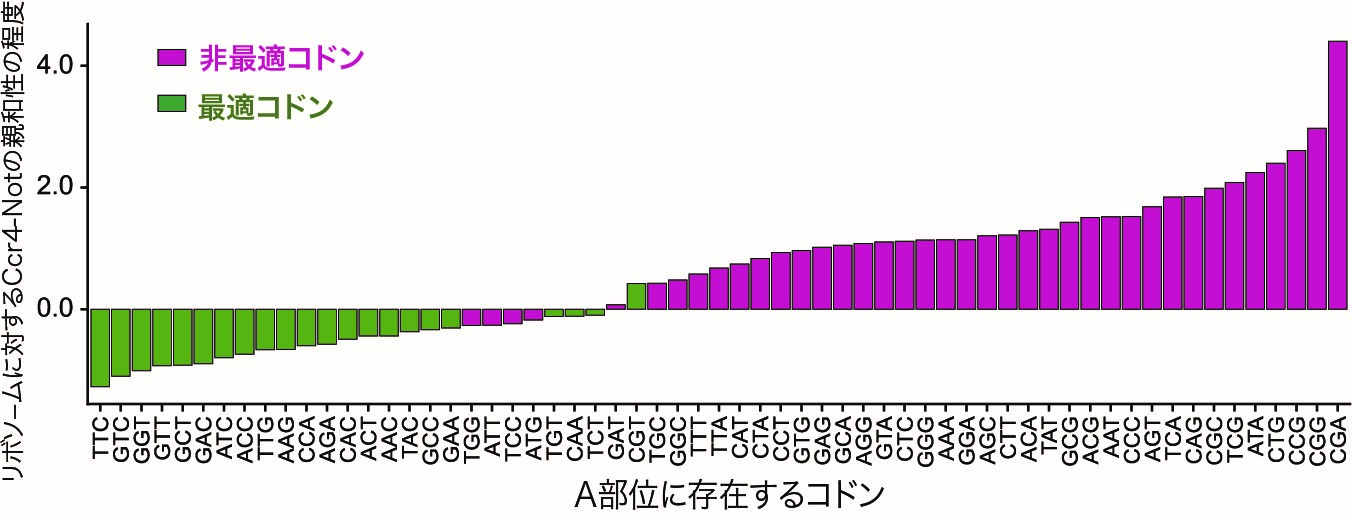
図1 Ccr4-Notとリボソームの親和性は最適コドンに強い逆相関を示す。
縦軸はリボソームに対するCcr4-Not複合体の親和性を、横軸はリボソームのA部位のコドンを示す。非最適コドン(赤)を含むリボソームはCcr4-Not複合体に高い親和性を示し、最適コドン(緑)を含むリボソームは低い親和性を示した。従って、Ccr4-Not複合体が結合するリボソームのA部位には、より最適度の低いコドンが存在することが明らかになった。
問い合わせ先
(研究に関すること)
東北大学大学院薬学研究科:稲田 利文教授
電話:022-795-6874
E-mail:toshifumi.inada.a3*tohoku.ac.jp(*を@に置き換えてください)
(東北大学に関すること)
東北大学大学院薬学研究科事務:星野公太郎
電話:022-795-6801
E-mail:kotaro.hoshino.d2*tohoku.ac.jp(*を@に置き換えてください)
