2022年 | プレスリリース・研究成果
ヒトゲノム複製におけるDNAポリメラーゼ間の分業と複製開始領域の同定 -ゲノム安定性とDNA複製機構の関わり合い-
【本学研究者情報】
〇学際科学フロンティア研究所 助教(研究開始当時) 大学保一
研究室ウェブサイト
〇生命科学研究科 教授 東谷篤志
研究室ウェブサイト
【発表のポイント】
- ヒト培養細胞を用いて、全ゲノムにわたり特定のDNAポリメラーゼが機能する領域を解析する実験方法Polymerase usage sequencing(Pu-seq)を確立しました
- DNAポリメラーゼのゲノムプロファイルを利用して、複製が開始される領域を今までにない精度で予測することに成功しました
- 今後は、本研究で確立されたPu-seq実験の応用により、がん細胞に特異的なDNA複製の仕組みが明らかになることが期待されます
【概要】
ヒトではDNA合成反応に働く酵素(DNAポリメラーゼ)が17種類存在し、それらのDNA合成反応の効率・正確性はそれぞれ異なり、ゲノム複製におけるポリメラーゼ間の分業は、ゲノム情報の安定性を決める主な要因です。特に、がん細胞においては、多くのDNAポリメラーゼ遺伝子に変異が生じ、DNAポリメラーゼの使われ方が大きく変化することが報告されていますが、その実態は明らかになっていません。我々は、ヒト培養細胞を使用して、全ゲノムにわたりDNAポリメーラの機能を解析する方法Polymerase usage sequencing(Pu-seq)法を開発し、主要なDNAポリメラーゼと言われていたPolε(イプシロン)とPolα(アルファ)それぞれが主にリーディング鎖・ラギング鎖合成に関与することを明らかにしました。また、これらのポリメラーゼのプロファイルを組み合わせた解析から、ゲノム上に多数存在する複製開始領域を今までにない精度で予測することにも成功しました。今後は、本研究で開発されたPu-seq実験によって、がん化などによる細胞の状態変化によって生じるゲノム複製機構の変遷を明らかにし、ヒトなどの大きなゲノムを持つ生物が潜在的に有するDNA複製の柔軟性(flexibility)、また、それに伴う脆弱性(fragility)の全容解明に向け、研究を大きく進めていく予定です。
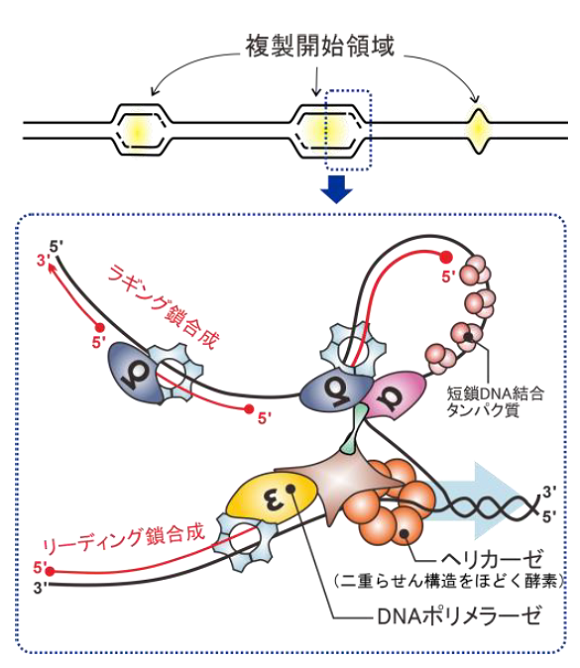
図1. DNA複製フォークの構造
ヘリカーゼがほどいた一本鎖DNA上でPolεがリーディング鎖、PolαとPolδラギング鎖合成を担う
問い合わせ先
(研究に関して)
公益財団法人がん研究会 がんゲノム動態プロジェクト
プロジェクトリーダー 大学保一
(研究開始当時 東北大学学際フロンティア研究所 助教)
電話: 03-6426-0435
E-mail:yasukazu.daigaku*jfcr.or.jp(*を@に置き換えてください)
(報道に関して)
東北大学学際科学フロンティア研究所
特任准教授 藤原英明
電話: 022-795-5259
E-mail:hideaki*fris.tohoku.ac.jp(*を@に置き換えてください)

![]()
東北大学は持続可能な開発目標(SDGs)を支援しています
